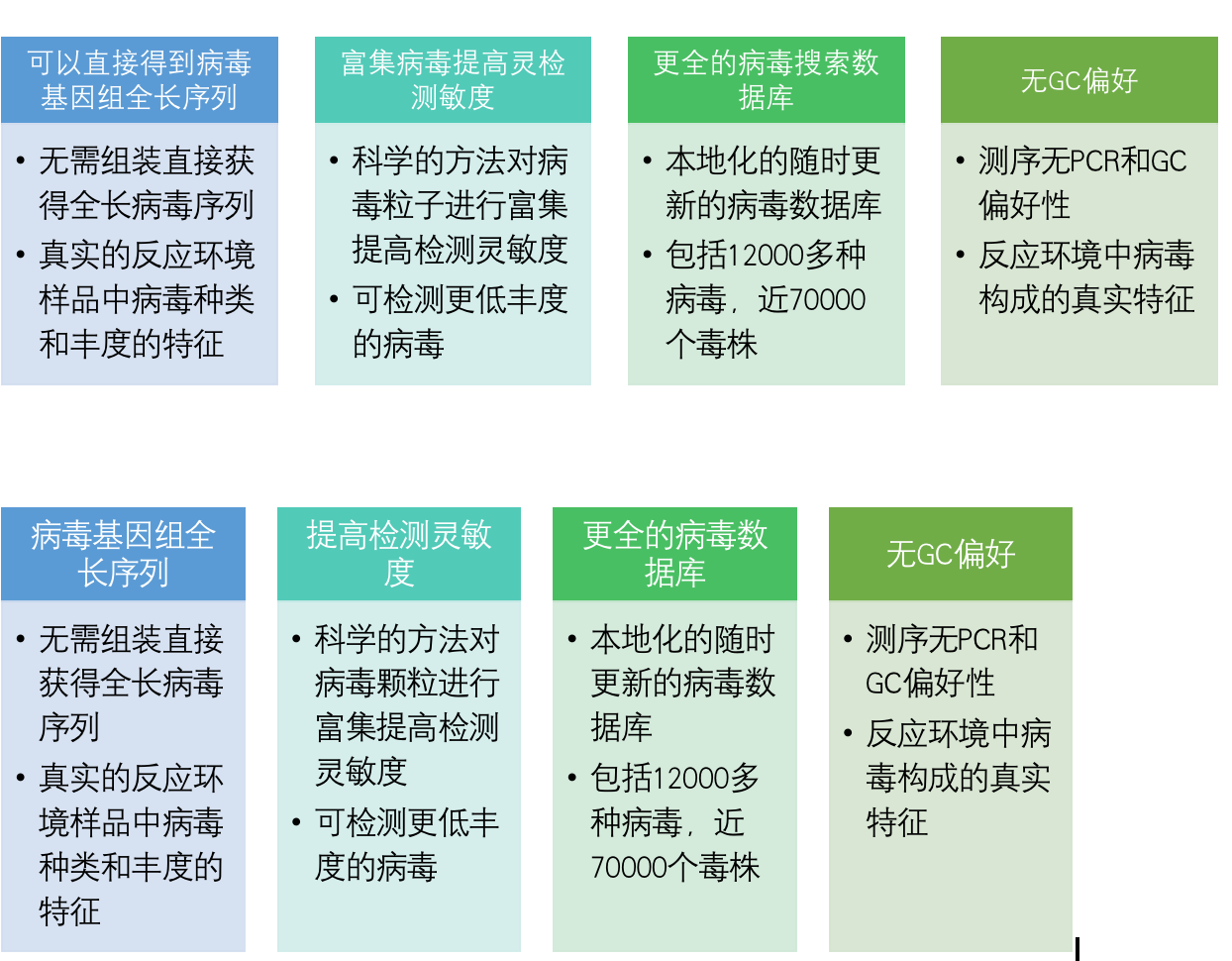
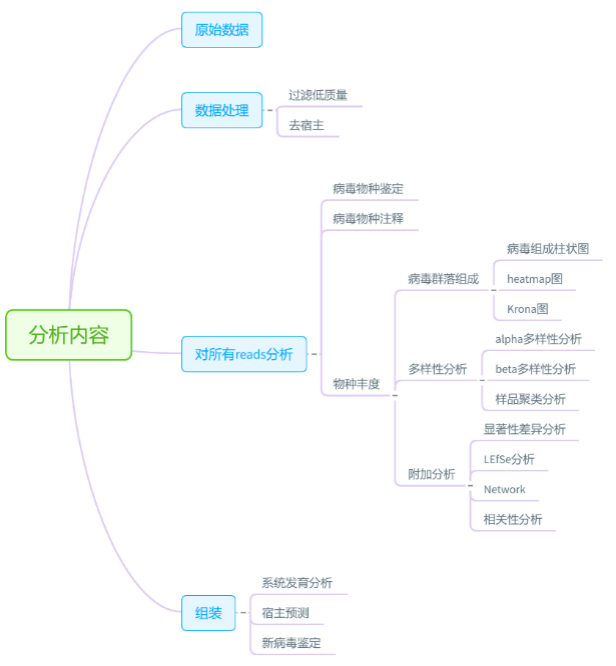
参考案例
无需组装,使用Nanopore测序从自然群体中识别完整的病毒基因组(IF= 11.20)
病毒是地球上最丰富的生物体,在宿主生态学、进化和基因水平转移中起着关键作用。由于病毒群体固有的遗传复杂性,很难从自然微生物组中获得完整的病毒基因组。该研究开发了一种基于Nanopore测序无需组装的检测方法,直接从环境样品中识别完整的病毒基因组序列。
材料与方法
采集3个不同深度的海水样品,经过过滤之后提取;使用GridION和Illumina测序;
利用大多数双链DNA尾噬菌体具有直接末端重复序列(DTRs)这一特征,对Nanopore reads进行过滤,对reads进行分bins,分别使用长reads和短reads对病毒基因组纠错。
研究结果
1. 模拟宏病毒组验证分析流程,结果显示,准确率≥99.67%,覆盖率≥99.86%。
2. 加入10ng λ噬菌体DNA作为内参进一步验证,获得的48,510bp基因组与参考基因组同源性为99.92%。
3. 该流程一共获得1864个高质量的基因组草图,水下25米、117米和250米的样品分别获得566、93和1205个病毒基因组(AFVG)。
4. 所有AFVG(1864个)均被VirSorter识别为病毒;AFVG注释基因与病毒数据库中病毒基因有很高的序列相似性。
5. AFVG的分类组成与已知的同一海洋区域的微生物群落以及浮游微生物宿主一致。
6. 该研究首次证明,PICI基因组串联重复序列可能被包装在“wild”噬菌体颗粒中,串联重复的大小反映了噬菌体的基因组大小。